�ό`���� tRNA�i2009.03.04�j
�Íۂł͂����� tRNA �̈�`�q�ɃC���g�������܂܂�Ă��܂��B ���ɂ� 2 �̓Ɨ������]�ʎY���Ƃ��č�������C1 �� tRNA �ɑg�ݏオ�� split tRNA ���m���Ă��܂������C Fujishima et al. (2009) �͂�͂�Íۂɂ����āCtRNA �� 3 �̓Ɨ������]�ʎY������g�ݏオ�� "tri-split tRNA" �����܂����B
����܂� split tRNA �͒��D�M���Ŋ��̌Íۂł��� "Nanoarchaeum equitans" �ł̂ݒm���Ă��܂����B "N. equitans" �͌Íۂ̒��ł��ŏ��̃Q�m���������Ƃ���C split tRNA �̓Q�m���̏��^���ƊW������Ƃ��v���܂������C���҂�͂��̉\���������邽�߁C ���̌Íۂ̃Q�m���ɂ����āC�������ꂽ tRNA ��T�����܂����B���҂�͓��ɒ��D�M���̌Íۂ� 1 ��C Caldivirga maquilingensis�i�N�����A�[�P�I�^��e�����v���e�E�X�ځj�̃Q�m���ɒ��ڂ��܂����B C. maquilingensis �͊��ɃQ�m������J����Ă��܂����C����܂ŕK�v�� tRNA �̂��� 6 �킪�������ł����B �����Œ��҂�͎��g�ŊJ������ tRNA �̗\���v���O�����Ȃǂ����p���Ė������� tRNA ��T�����܂����B
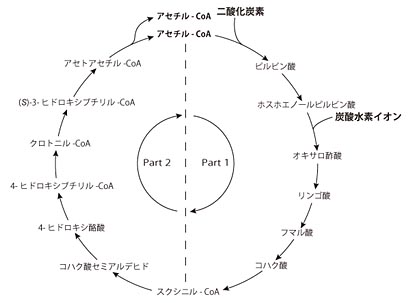
�܂����߂̒T���� 4 �� split tRNA ����������itRNA Gly (CCC)�CtRNAAla (CGC) �� (TGC)�C����� tRNAGlu (TTC)�j�C����ɒT���������� tRNAGly (TCC) �� (GCC) �ɑΉ����� 3 �� RNA �f�Ђ���������܂����B���� 3 �̒f�Ђ� tRNAGly (CCC) �̒f�Ђ� 1 �Ƃ��g�ݍ��킹�� 2 ��ނ́C 3 �f�Ђ���Ȃ� tRNA �ɑg�ݏオ��Ɛ��肳��܂����i���}�j�B
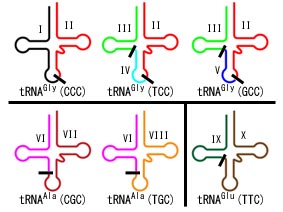 |
| Split tRNA �� tri-split tRNA�i���[�}�����͒f�Ђ̔ԍ��j |
�����̕����^�� tRNA �̊e��`�q�́C�אڂ���̈悪 bulge-helix-bulge�iBHB�j���`�[�t�i�܂��͂���ɗނ���\���j �������Ă���C����� C. maquilingensis �̃X�v���C�V���O�E�G���h�k�N���A�[�[�ɂ���Đؒf�E �A�������ƍl�����܂����i���}�j�B�܂��e�f�Ј�`�q�̓v�����[�^�Ɛ��肳���z��������Ă���C ���ۂɓ]�ʂ���Ă��邱�Ƃ��m�F����܂����B����ɂ��ꂼ��� tRNA �f�Ђ̌����itri-split tRMA �̏ꍇ�CIII+IV�CIV+II�C III+V�CV+II �����̌������j�������I�Ɋm�F����C�������� tRNA ���@�\���邱�Ƃ��F�߂��܂����B
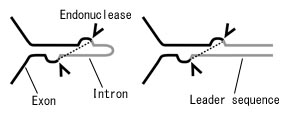 |
| Bulge-helix-bulge�iBHB�j���`�[�t |
�Ō�ɁC�e�����v���e�E�X�ڂ̃Q�m���z��Ƃ̔�r����C�����^�� tRNA �� leader sequences �͑���� tRNA �̃C���g�����Ƒ����ł���Ǝw�E����Ă��܂��B�������҂�̓C���g�������܂� tRNA ���番���^�� tRNA ���Q�m���z��̕ψقɂ���Đi�������Ƃ͍l���ɂ����Ǝ咣���Ă��܂��itRNA ���@�\���ێ������܂܈�`�q���f�Љ����C �㔼�������v�����[�^�[�z���V���Ɋl������͍̂���Ƃ̂��Ɓj�B ����Ńv���Z�V���O���Ă���r���� tRNA �f�Ђ��C�t�]�ʂɂ���ăQ�m�����ɑ}�����ꂽ�\�����c�_���Ă��܂��B
�Q�m�����̕ψقŃC���g������������ tRNA ���番���^�� tRNA ���o�������\�����\���ɂ���Ƃ͎v���܂��� �i�Ⴆ�Ώd�������C���g�������܂� tRNA �̕Е����C���g�����̌㔼�����ƌ㔼�̃G�L�\���������C ��ɂ�������̃R�s�[�ōŏ��̃G�L�\���ƃC���g�����̑O������������������Csplit tRNA ����������G���}�j�C ���ۂ̐i���ߒ��𖾂炩�ɂ���ɂ́C�߉��ȌÍۂ̃Q�m���Ԃ� tRNA �̍\�����r���邱�Ƃ����҂���܂��B �Ȃ��C���҂�� tRNA �̐i������Ղ��邽�߂Ɋ����Ɋ܂܂��Íۂ̃Q�m���i���^�Q�m���j�z��� tRNA ����͂��邱�Ƃ��Ă��Ă��܂��B
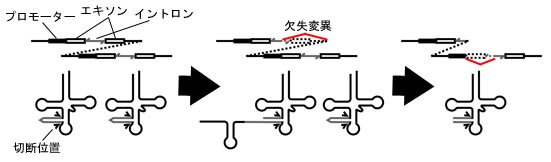 |
| �����^�� tRNA �̋N���Ɋւ����։��� |
Fujishima, K. et al. Tri-split tRNA is a transfer RNA made from 3 transcripts that provides isight into the evolution of fragmented tRNAs in archaea. Proc. Natl. Acad. Sci. USA 106, 2683-2687 (2009).