まずファイルを開きます。ファイルを seaview.exe に放り込んでも開けますが,予め seaview.exe を開き, File → Open としてファイルを開きましょう。
ちなみに SeaView は Clustal 形式(*.aln)以外に Mase 形式(*.mase),Phylip 形式(*.phy),MSF 形式(*.msf), FASTA 形式(*.fst)および NEXUS 形式(*.nxs)のファイルを読み込むことができます。File → Open Clustal などとすることで, 各形式の拡張子を持ったファイルを選んで開くこともできます。
正しくファイルが開ければ,中央部分にアラインメントが表示されるでしょう。
最初にアラインメントのギャップの位置を修正してみましょう。
図 1 の様な場合,"TC" という並びが全ての種で相同であると思われるため,"TC" が縦に並ぶように左右のギャップを調節したいと思います。
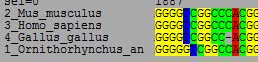 |
| 図 1 |
マウスで操作したい位置をクリックして,カーソルをその位置に持って行きましょう(カーソルは矢印キーでも移動できます)。 そこで "-"(ハイフン)キーを押すと,カーソルの位置にギャップ("-" で表記される)が挿入されます。 逆に "Back Space" または "Delete" キーを押すとカーソルのすぐ左に位置するギャップが削除されます。 これらのキーを用いてアラインメントのずれを修正しましょう(図 2;カーソルの位置の塩基が黒くなっています)。
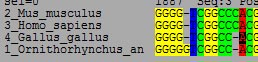 |
| 図 2 |
次に,アラインメントの末端を整えてみましょう。
集めた配列によっては末端がそろっていないことはしばしばあります(例えば,図 3)。
 |
| 図 3 |
しかし一部の配列にのみギャップがある場合,系統解析に悪影響を及ぼす可能性があります。そこで末端部分を切り取り, 全ての配列が揃っているサイトからアラインメントが始まるように編集しましょう(一部の配列にギャップがあっても, 他の配列に十分な情報が含まれている場合もあるため,常に末端を揃えるべきとは限らないので注意)。
ギャップ以外の配列を削るためには,配列の編集を許可する必要があります。これはメニューの Props にある "Allow seq. edition" にチェックを入れることで可能になります。 "Allow seq. edition" がオンになっている場合,ギャップも含めた全ての塩基が削除可能になり, また塩基配列の書き込みもできるようになります(カーソル部分に文字を打ち込むと,文字が挿入されます)。
末端配列を削除する時,一行ずつ削除していくのは非効率的です。また一端アラインメントをずらす形になるため, どこから削ればいいのか分からなくなってしまうかもしれません。そこで便利なのが配列(種)を選択する機能です。 マウスを用いて配列名をクリックすると,その配列名が黒く反転します(もう一度クリックすると解除されます)。 この状態では黒く反転した配列が選択されていることになります。同様に複数,あるいは全ての配列を選択することも可能です (いずれかの配列名をダブルクリックすると全ての配列が選択されます。同様に右クリックまたは Shift + クリックで全ての選択が解除されます)。
さて,複数の配列が選択されている状態で,カーソルを選択されている配列に置いてギャップの挿入や削除, 配列の削除を行うと,選択されている全ての配列に変更が反映されます。例えば全ての配列を選択している状態で "Back Space" または "Delete" キーを押すと,カーソルのすぐ左の縦一列が削除されます(注:"Allow seq. edition" がオフになっている場合は,すぐ左の列のギャップのみが削除されます)。
この機能を用いると列ごと削除が行えるため,末端を簡単に揃えられます(図 4;左端を揃える場合には, 揃っている最初の列にカーソルを置き,"Back Space" を押し続ければよい)。
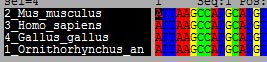 |
| 図 4 |
注:このプログラムには「元に戻す」の機能がありません。削除は慎重に行いましょう。 不安がある場合は,削除作業を行う前に一度保存しておくと安心です(保存は次の項を参照)。
両端を揃えたら,一度編集したアラインメントを保存しましょう。
File → Save または File → Save as... とします。
Save はファイルに上書きし,Save as... では別名で保存できます。 "Save" は途中で Props → Save Format などで形式を一度でも変更した場合,選択できなくなります。"Save as..." の場合には, 保存形式が Mase 形式,Phylip 形式,Clustal 形式,MSF 形式,FASTA 形式および NEXUS 形式から選択できます。
保存形式は今後の用途によって選ぶべきですが,ClustalX に戻すのであれば Clustal 形式か FASTA 形式がよいでしょう。
注:一部の NEXUS 形式のファイルから FASTA 形式への保存を試みるとプログラムが停止するバグがありましたが, 2008年03月09日,Dr. Manolo Gouy に修正していただきました。
最後に,範囲(Site sets)を指定するという,やや複雑な方法を紹介します。
系統解析の際に,アラインメントの一部だけ(例えばタンパク質の機能ドメインだけ)を使用したいと言う場合や, 逆に一部だけ(例えばアラインメントが曖昧な領域)を省きたいという場合が存在します。例えば図 5 ではアラインメントにやや不安がある領域を示しています(アラインメント上側の黒線)。
 |
| 図 5 |
SeaView にはこのような領域を指定する(そして後で除外する)ための機能が備わっています。それが Site sets です。 Site sets は配列の編集なしに設定可能で,指定された領域を選択的に保存することができます。 つまり系統解析に用いたい部分に Site sets を指定すると,配列を編集しないで保存することもできますし(Site sets の指定も保存される),系統解析に用いたい部分だけを独立のファイルに保存することもできるのです。
以下に具体的な手順を示します。
Sites → Create set として set の名前を入力します(**** domain や unambiguously aligned sites など)。 すると画面に表示されているアラインメントの一番下に,入力した名前の行が出現します(初期設定では白抜き文字)。 初めはその行の「配列」は全て "-" になっているはずです。
この行をクリックまたはドラッグすると,その範囲が "X" に変わります。後の解析に用いたい領域を "X" で指定してください(図 6;指定から外れた部分がやや灰色がかって表示されます)。 なお,この "X" は再度クリックすると "-" に戻ります(注:"X" と "-" が接している時には, クリックしただけでは "X" から "-" への変更は出来ません。隣の "X" にドラッグしたときにのみ "-" に戻ります)。
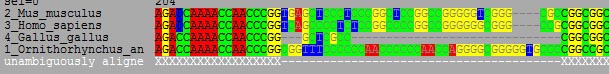
図 6 Site sets の指定は Sites → Save set で確定します。確定しないまま Sites → Hide set で表示を隠すと, または他の set(同時に複数の sets が指定できます)に表示を切り替えると,指定がリセットされてしまいます。 従ってこまめに確定することをお勧めします。
(確定した)Site sets を含めた情報をファイルに保存します。この際,Mase 形式か NEXUS 形式でしか Site sets の情報は保存できません。この過程は必須ではありませんが,系統解析に用いた領域を記録しておく意味があります。
File → Save selection として,Site sets で指定した領域だけを切り出して保存します。 ファイル名(初期値では regions となっている)と形式を指定して保存してください。なお,特定の配列が選択されている場合には, その配列の指定された領域だけが保存されます。
保存したファイルを再度 SeaView などで開いて確認します。"X" で指定しなかった領域が削除されているはずです (図 7;矢印が削除された部分)。

図 7